神経画像を用いた新規診断アルゴリズム(自動診断システム)の開発
杉原 玄一
ロンドン大学精神医学研究所 リサーチワーカー
(Section of Neuroimaging,
Department of Psychological Medicine and Psychiatry
Institute of Psychiatry, King's College London)
1.はじめに
本ファイル群は、構造MRI画像により自閉症の診断を行うことを目標に開発されたプログラムであ る。本プログラムは研究目的で使用されることを前提に作成されており、現時点では臨床場面において 使用されることを想定していない。
2.内容
MRI_Data: Statistical Parametric Mapping (SPM5) softwareによって前処理された灰白質の構造MRIデータ。 Analyze形式。自閉症群13名、健常対照群13名。sm0wc11*.img/hdrが自閉症群。sm0wc12*.img/hdrが 健常対照群。
Preprocessing: GE社製3T-MRI によって撮像された構造MRIデータの画像変換、前処理するためのスクリ プト。MATLAB (MathWorks社製、ヴァージョン7以上)、SPM5
(http://www.fil.ion.ucl.ac.uk/spm/software/spm5/)、VBM5.1 (http://dbm.neuro.uni-jena.de/vbm/vbm5-for-spm5/) がイ ンストールされている環境が必要。
dicomimport.m: GE 社製3T-MRI によって撮像された構造MRI生データ(DICOM形式)をAnalyze形式に変換 するためのスクリプト。
dicomimporttmp.mat: GE社製3T-MRI によって撮像された構造MRI生データ(DICOM形式)をAnalyze形式 に変換するためのjobファイル。
VBM_WriteAndEstimate.m: VBM5.1によりsegmentation、spatial normalisation を行うためのスクリプト。
VBM_WriteAndEstimatetmp.mat VBM5.1 によりsegmentation、spatial normalisationを行うためのjobファイル。
smooth.m: スムーシングのためのスクリプト。
smoothtemp.mat: スムーシングのためのjobファイル。
probid_v1.017: 機械学習を行うプログラム。MATLABがインストールされている環境が必要。
Results: 自閉症群13名、健常対照群13名を用いPROBIDにより解析した結果。
3.解析の流れ
GE社製3T-MRI によって撮像された構造MRIデータ(DICOM形式)をdicomimport.mによりSPM5を 起動しAnalyze形式に変換する。
各個人にAnalyze形式の脳画像を入れるフォルダ1つを作成する。フォルダは数字とし、自閉症群は 1002~10014、健常対照群は2001~20013とする。
VBM_WriteAndEstimate.mによりVBM5.1を起動しsegmentation、spatial normalisation を行う。
smooth.mによりSPMを起動し8mm full width at half maximum (FWHM) でスムーシングする。
上記前処理されたAnalyze形式の画像(sm0wc1*.img/hdr)を1つのフォルダにコピーする。
PROBIDを起動し機械学習を行う。
4.結果
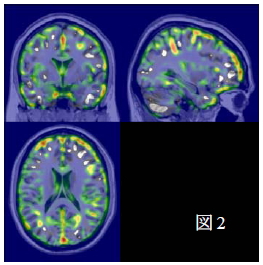
自閉症群13名、健常対照群13名の灰白質14MRI画像を用いた機械学習で感度69%、特異度77%で両群を判別することが可能であった(図1)。
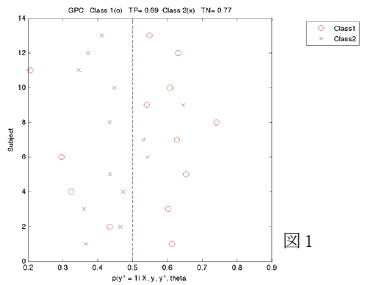
判別領域の分布パタンでは前頭葉を中心とした広範囲の皮質で自閉症群と健常対照群との違いが示された(図2)。